Une planification pré-procédurale complète, fiable et rapide pouvant être facilement intégrée au processus intra-procédural est d'une importance primordiale, qu'il s'agisse de délimiter des cibles anatomiques ou d'identifier la trajectoire optimale qui évite les organes vitaux environnants. Sous ce thème de recherche, nous développons de nouvelles approches en modélisation de forme statistique et en anatomie computationnelle basées sur l'apprentissage renforcé pour classifier, segmenter et enregistrer des images diagnostiques multimodales à haute résolution.
Développement d'une plateforme d'élastographie par ultrasons 3D pour l'analyse in vivo de la tension de la paroi de l'anévrisme de l'aorte abdominale
Hiver 2020: Hongliang Li (Postdoc)
L'anévrisme de l'aorte abdominale (AAA) est une dilatation anormale de l'aorte dans l'abdomen due à un affaiblissement de la paroi causé par l'athérosclérose. La rupture de l'AAA est la 13e cause de décès en Amérique du Nord. Bien que les indications pour une intervention de rupture sont basées sur le diamètre maximal des AAA (5 cm), 23% des AAA rompus mesurent moins de 5 cm et dans les grands AAA, le taux de rupture pourrait être inférieur aux attentes. Cette seule indication induirait une sous-estimation ou une surestimation du risque de rupture.
L'objectif du projet est d'utiliser l'imagerie ultrasons (US) en 3-D pour créer une cartographie 3-D de la souche AAA et enregistrer la cartographie de la souche sur un scanner monophasé pour améliorer la caractérisation de la vulnérabilité AAA, qui pourrait être un nouveau biomarqueur de risque pour prédire la croissance de l'AAA et la rupture.
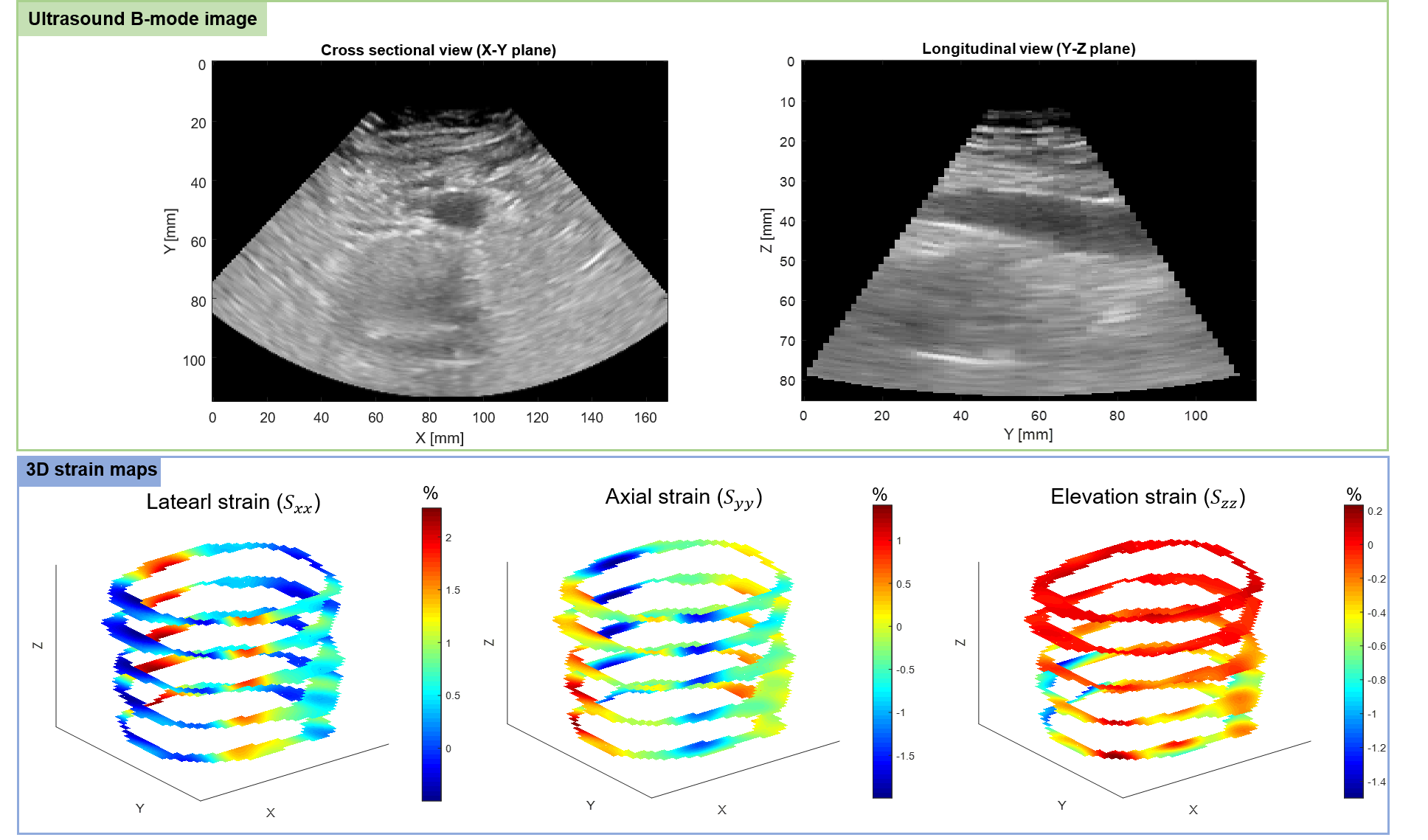
Soumission: IUS 2020
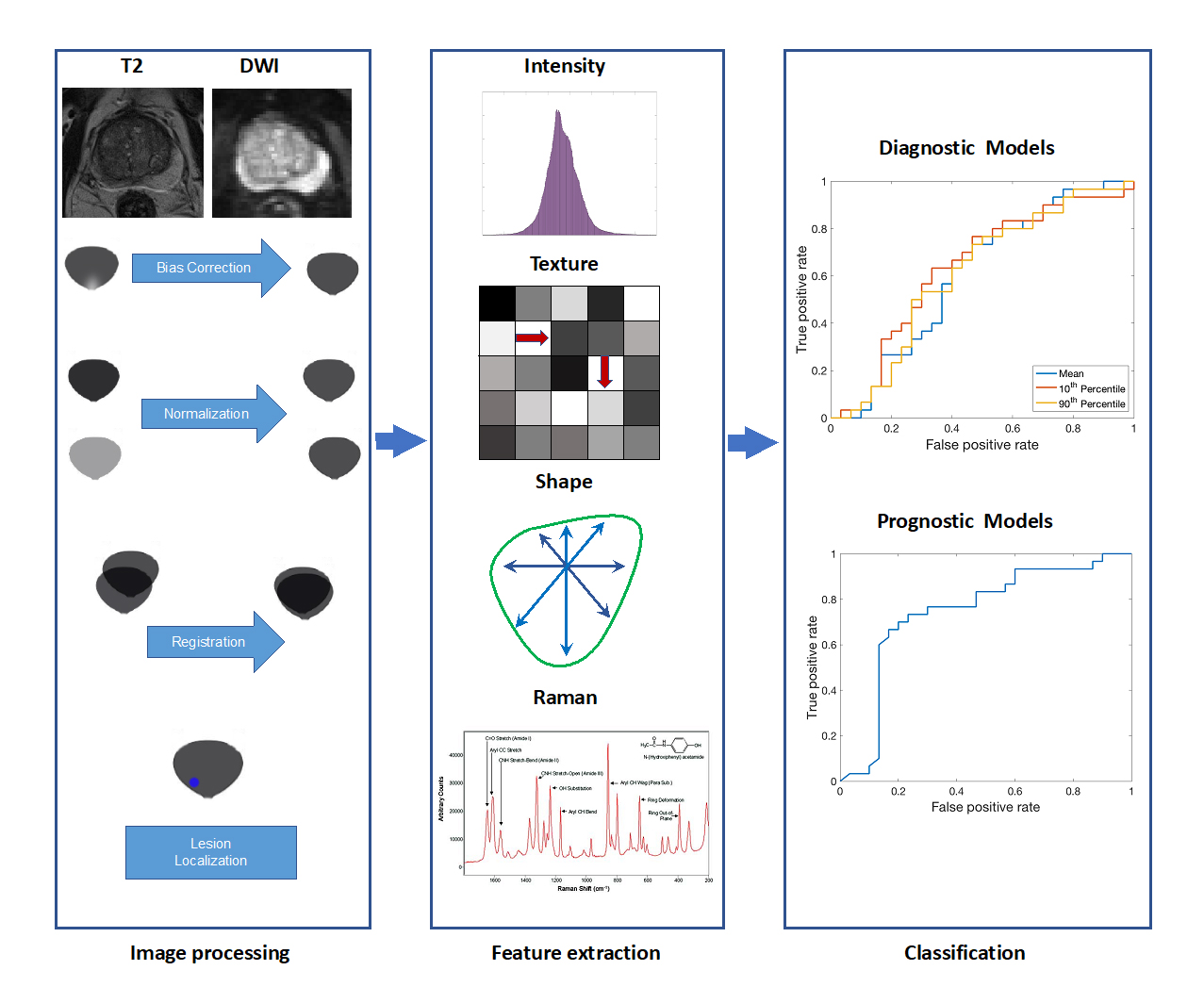
Rôle des biomarqueurs de résonance magnétique quantitative dans les interventions ciblant les tumeurs de la prostate
Stephanie Alley (Doctorat)
L'imagerie par résonance magnétique multiparamétrique (IRMmp) s'est révélée être un excellent outil de diagnostic, de stadification, de planification, de traitement et de surveillance du cancer de la prostate en raison de sa capacité à décrire le contraste des tissus mous à haute résolution. Malgré les progrès réalisés dans ce domaine, des divergences apparaissent souvent entre les résultats des études en raison d’un manque de reproductibilité dans les analyses par IRMmp. L’objectif de ce travail est de développer un processus qui inclura des méthodes de traitement et d’analyse liées à l’IRMmp de la prostate et qui permettra d’identifier des biomarqueurs de résonance magnétiques robustes et fiables pouvant être utilisés pour cibler efficacement les tumeurs de manière interventionnelle.
Aborder la variabilité multi-évaluateurs de la segmentation manuelle des lésions dans les images médicales et son impact sur les modèles de segmentation d'apprentissage en profondeur
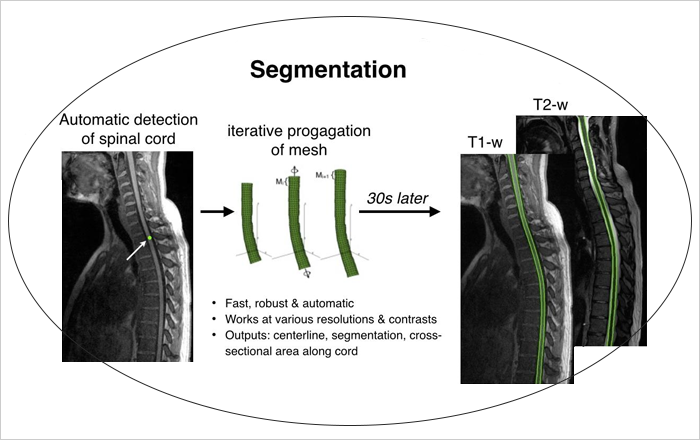
Automne 2021: Zeinab Abboud (Doctorat)
La segmentation de la moelle épinière a une grande importance pour de nombreuses applications, allant du planning de chirurgie au diagnostic/pronostic de maladies neurodégénératives. Il n’existe cependant que peu de techniques segmentant automatiquement la moelle épinière pour différents types de contraste IRM. L’objectif principal de ce projet est de développer une méthode de segmentation automatique de la moelle épinière pour des images IRM pondérées en T1 et en T2, qui soit capable de gérer la courbure variable de la moelle épinière et les manques de contrastes et artéfacts éventuels. Cette méthode se basera sur la propagation multi-résolution d’un modèle déformable, couplée à des mécanismes d’adaptation au contraste local. Un module indépendant permettra la détection de la moelle épinière dans un volume IRM et fournira à la méthode la position et orientation approximative de la moelle épinière. Couplée avec un algorithme d’identification des niveaux vertébraux, la méthode développée fournira des mesures quantitatives de la moelle épinière (e.g. aire de section transverse, volume) et permettra une comparaison inter- et intra-patient pour de larges cohortes. Cette méthode sera disponible gratuitement en ligne sur http://sourceforge.net/projects/spinalcordtoolbox/.
L’épilepsie bénigne à pointes centrotemporales (EBCT) est l'un des syndromes épileptiques de l'enfant les plus courants et se produit chez les enfants âgés de trois (3) à 13 ans. Initialement, l’EBCT a été considérée comme bénigne, mais récemment certaines études ont montré des déficits cognitifs et comportementaux qui peuvent persister même après la rémission. Des études de neuro-imagerie récentes ont montré le lien entre ces déficits et les dysfonctionnements cognitifs dans des structures spécifiques du cerveau ce qui montre la possibilité de modifications neuroanatomiques dans ces régions du cerveau. Dans ce projet de recherche, notre but est de proposer un cadre d'analyse morphologique automatique EBCT pour détecter les altérations neuroanatomiques subtiles chez les enfants avec EBCT par rapport à des témoins normaux. À cette fin, nous développons un processus de co-recalage et co-segmentation par groupe itératif, qui permet la segmentation automatique des structures sous-corticales dans plusieurs images. Ensuite, nous concevons un cadre pour l'adaptation des maillages de surface sous-corticales 3D et d'enquêter sur les différences structurelles de groupe entre deux populations de surfaces, c'est-à-dire, des sujets sains et pathologiques. Enfin, nous proposons une méthodologie pour évaluer l'association entre les altérations morphologiques et la cognition.
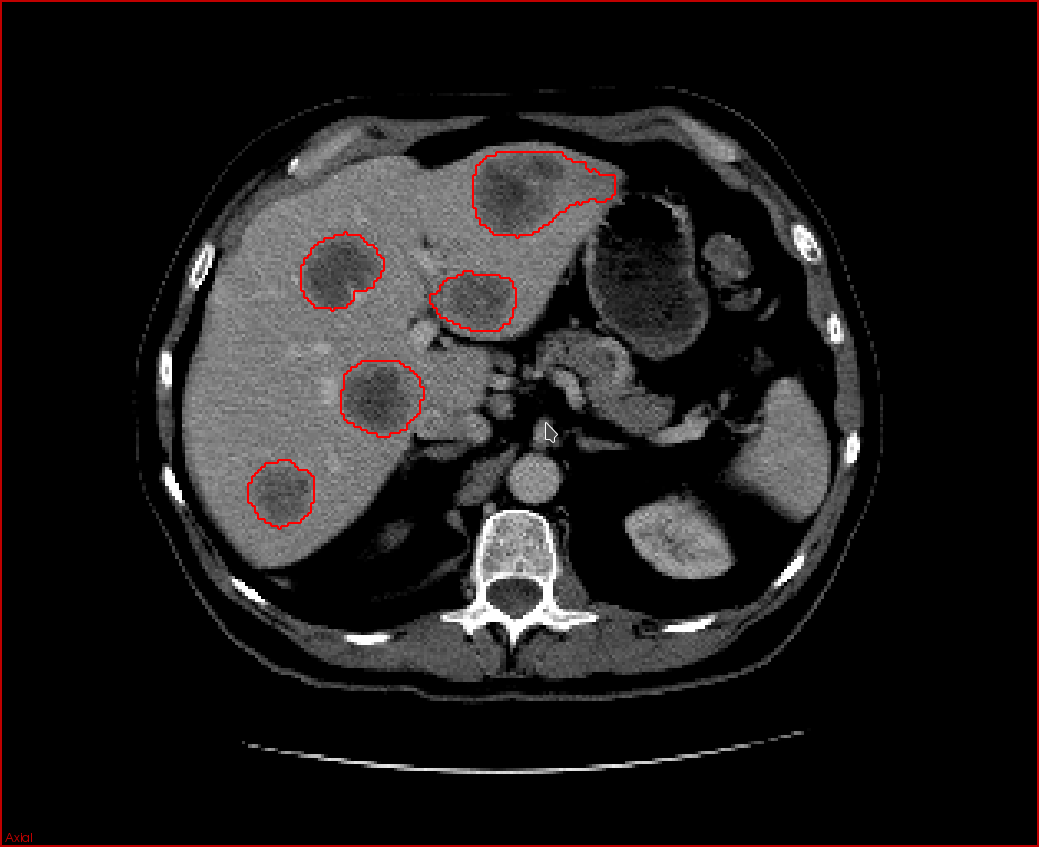
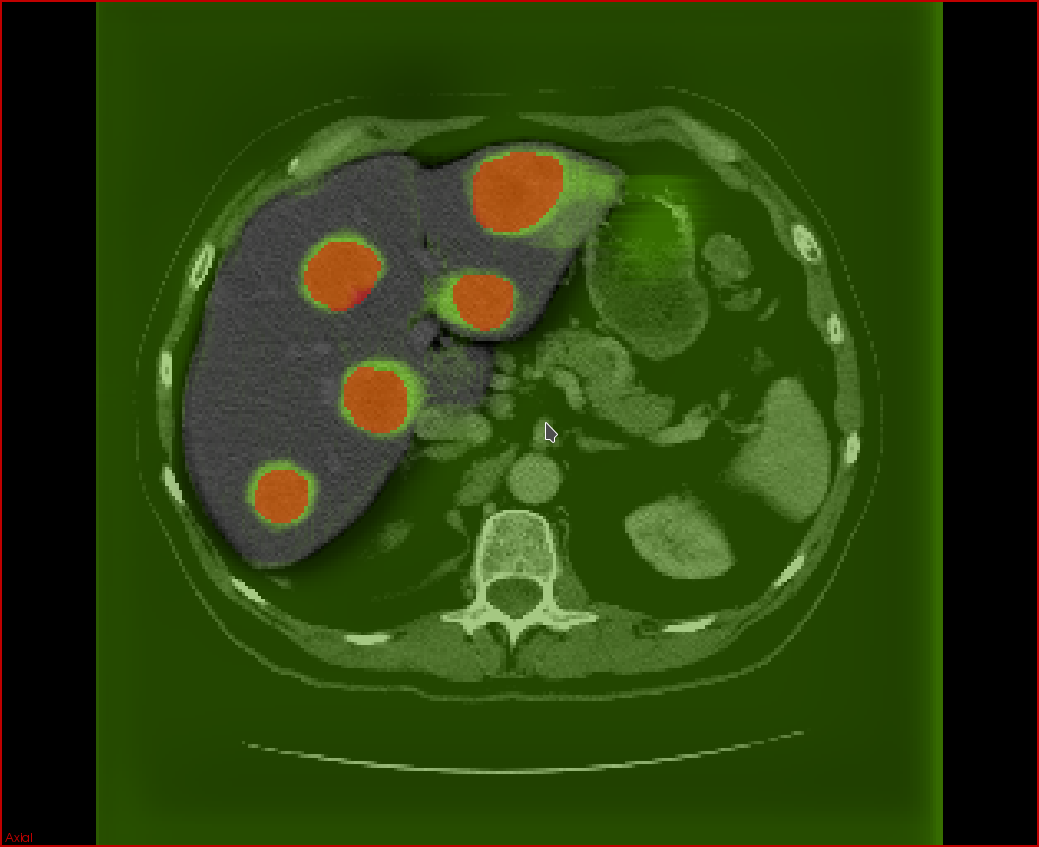
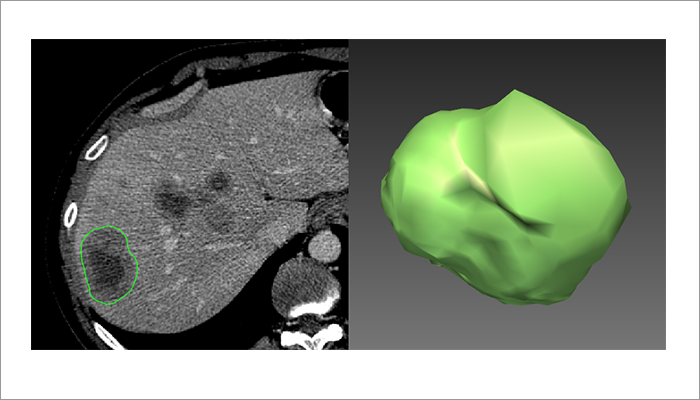
Les tumeurs secondaires du foie (métastases) ainsi que les tumeurs hépatiques primaires (carcinome hépatocellulaire) sont les causes les plus fréquentes de mortalité liée au cancer en Amérique du Nord. Actuellement, il n'y a pas suffisamment d'automatisation dans le traitement des scans 3D des tumeurs à partir des modalités d'imagerie telles la tomodensitométrie (TDM) et l'imagerie par résonance magnétique (IRM). En effet, il y a notamment beaucoup de place pour le développement de l'automatisation de la classification des tissus, de la détection des lésions, et de la segmentation de la lésion et des organes. Dans le contexte du diagnostic du cancer, de tels outils servent à améliorer le diagnostic des tumeurs cancéreuses assisté par ordinateur (CADx).
Le but de ce projet est d'aider les radiologistes à réduire le taux de faux négatifs ainsi que le temps nécessaire pour offrir un diagnostic et un suivi précis. L'objectif est de développer des outils basés sur les techniques d'apprentissage profond pour un logiciel CADx utilisé pour le cancer qui peut être bien intégré dans un PACS ("Picture Archiving Communication System") et un RIS ("Radiology Information System").
La segmentation automatique des lésions permettra une estimation précise du volume et de la perfusion de la tumeur, aidant à estimer la charge tumorale et à évaluer les critères de réponse pour les tumeurs solides. Actuellement, ces évaluations sont obtenues en utilisant des mesures 2D, telles que la longueur et la largeur des tumeurs. Les modèles de réseaux de neurones profonds requièrent des quantités importantes de données pour former des modèles performants, ce qui représente un défi en raison de la rareté des sujets pouvant présenter des types de tumeurs d'intérêt et de la difficulté d'étiqueter un grand nombre d'images médicales pour la formation. En utilisant des données étiquetées manuellement par des collaborateurs cliniques, nous prévoyons développer des modèles qui permettront de détecter et de segmenter les tumeurs dans un foie segmenté à partir d'images CT et IRM. Le développement de ces modèles permettra l'amélioration de la précision ainsi qu'une variabilité réduite et ce, avec une intervention humaine minimale. Cela a le potentiel d'élargir l'utilisation de l'imagerie médicale pour le dépistage de la tumeur, le diagnostic et le suivi, et donc d'améliorer les soins aux patients.
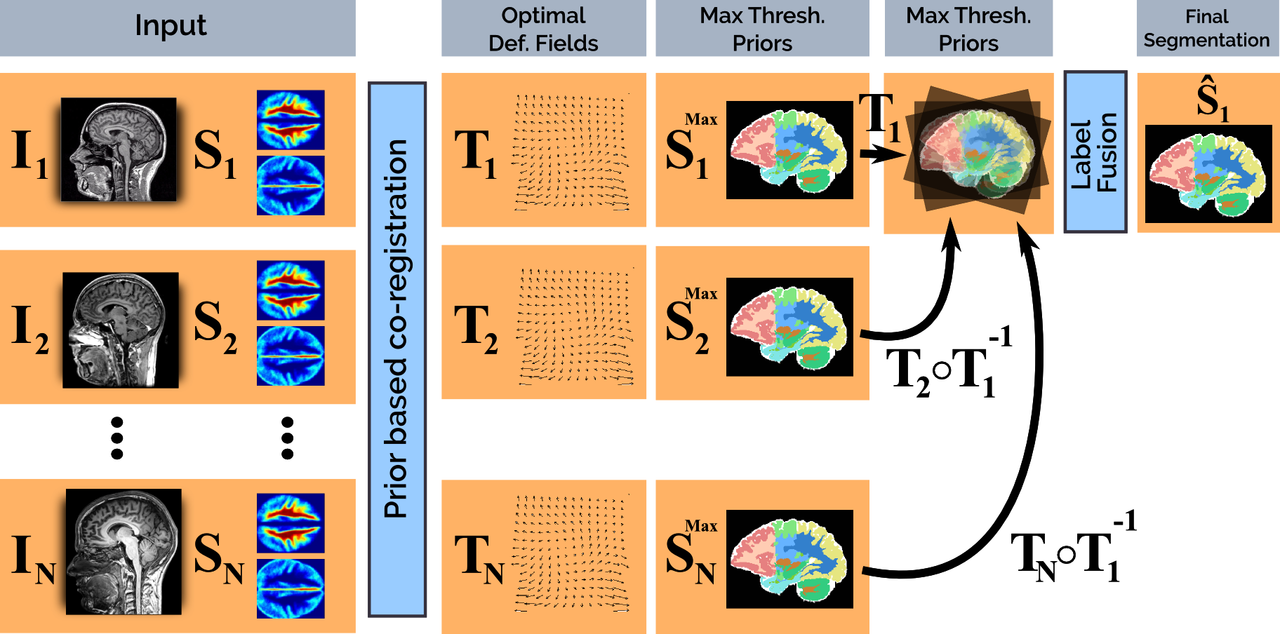
L’IRM est la méthode d’imagerie standard pour le dépistage de troubles neurologiques avec des acquisitions du cerveau. Pour les nouveau-nés, les nouvelles sondes à ultrasons (US) peuvent acquérir des images 3D quasi instantanément et sont faciles à utiliser. Celles-ci permettent de réaliser une acquisition volumétrique de manière quasi instantanée. Dans un premier temps, une étude comparative d'images IRM et 3D US acquises pour chaque patient est réalisée pour comparer le volume cérébral US et IRM ainsi que le volume des ventricules. Par la suite, un recalage d’images est réalisé entre l’US et un atlas IRM afin de faire correspondre les régions d’intérêts. Puis, une segmentation est réalisée afin d’extraire les ventricules ou autres régions d’intérêts. Cette approche permettra d’extraire des voluimes en 3D du cerveau avec des images d’US 3D au lieu de l’IRM réduisant la complexité et le temps requis.
En clinique, les techniques de segmentation manuelles sont utilisées sur les images CT pour le diagnostic de tumeurs hépatiques. Des recherches récentes sur les méthodes de segmentation automatisées ont été réalisées en utilisant l’intelligence artificielle. Cependant, la prédiction et la segmentation des lésions hépatiques en utilisant des scans de suivi restent problématiques dues à de nombreux défis tels que l’évolution de la tumeur et la croissance des lésions. Le but de ce projet est de prédire ces changements à partir de scans de suivi en utilisant les développements récents en intelligence artificielle et en apprentissage profond.
Le point le plus important pour le succès d'un traitement de radiothérapie est de cibler précisément les tumeurs tout en épargnant les tissus sains du rayonnement ionisant. Cela demande une calibration précise des rayons, généralement effectuée en utilisant les images préopératoires. En recalant les images préopératoires de l'anatomie du patient sur sa position durant le traitement, il est possible de connaître la position des tumeurs. Cependant, lors d'une radiothérapie, le patient peut bouger et est libre de respirer. La position des tumeurs ne correspond donc plus à celle donnée par les images préopératoires recalées.
L'idée de ce projet est de prédire le mouvement des tumeurs à l’intérieur du foie pour ajuster la direction des rayons ionisants en temps réel afin de maximiser l'exposition des tumeurs en minimisant les dommages causés aux tissus sains.
Nous proposons de construire un modèle de mouvement à partir d'images IRM dynamiques de l'abdomen lors de plusieurs cycles respiratoires prisent sur un échantillon de patients. Ce modèle sera ensuite personnalisé sur les données de patients à traiter en utilisant des méthodes de « machine learning ».
La segmentation des tumeurs carcinomes du foie à partir d'images abdominales IRM ou CT est importante pour diagnostiquer et traiter le cancer du foie. Actuellement, cette segmentation est effectuée manuellement; cependant, la segmentation manuelle requiert beaucoup de temps et nécessite des connaissances spécialisées. L’automatisation du processus de segmentation est un domaine de recherche actif mais les méthodes disponibles n'atteignent pas une performance adéquate. L'objectif de ce projet est de développer un modèle afin d'accomplir la tâche difficile de segmenter automatiquement les tumeurs cancéreuses du foie à partir d'images IRM. La segmentation des tumeurs du foie est particulièrement complexe en raison de leur taille, de leur forme, de leur intensité et de leur texture très variables. Cette variabilité sera incorporée dans le modèle en utilisant l'apprentissage automatique. Un autre défi est la petite quantité des données d’entraînement disponibles en raison de la petite taille des ensembles des données disponibles pour un système d'imagerie donné. Compte tenu de ces défis, un classificateur approprié sera développé et combiné à un modèle de surface déformable afin d'affiner le résultat de la segmentation.
Le recalage de l'IRM pré-opératoire avec fluoroscopie à rayons X à bras en C est un processus complexe. Les progrès récents dans le domaine de l'apprentissage profond avec les réseaux antagonistes génératifs (RAG) ont montré des résultats prometteurs dans la synthèse des images du scan CT du cerveau en lien avec leur IRM pré-opératoire. Notre objectif final est le recalage automatisé des vertèbres entre les images d’IRM avec celles de la fluoroscopie à bras en C, avec celles obtenues par le scan CT, puisque le recalage automatisé des images obtenues par le scan CT et fluoroscopie à bras en C est plus facilement réalisable. Nous allons d'abord entraîner un RAG pour prédire les images CT des vertèbres en fonction de leur IRM, puis recaler automatiquement les images CT synthétisées avec la fluoroscopie à bras en C. La même transformation sera ensuite appliquée à l'IRM initiale pour obtenir un recalage final des images d’IRM avec celles de fluoroscopie à bras en C.
Le mouvement et la déformation du foie pendant la respiration sont des problèmes importants lors des procédures de ciblage tumoral telles que la radiothérapie : pour maximiser l'exposition des tissus néoplasiques et minimiser les dommages aux tissus sains, le point focal du faisceau de rayonnement doit suivre la position de la tumeur. Cependant, pendant ce type de traitement, le patient est libre de respirer et le mouvement de la tumeur hépatique doit en être compenser.
Le but de ce projet est de construire un modèle de mouvement du foie pendant la respiration qui prenne en compte les variations inter-cycliques pour prédire la position de la tumeur pendant la radiothérapie et permettre un meilleur ciblage de la tumeur. La méthode repose sur les methodes de réseaux de neurones profonds génératifs pour apprendre les motions des organes à partir de données populationnelles.
Le carcinome hépatocellulaire (CHC) est le sixième cancer le plus répandu, la deuxième cause de décès par cancer dans le monde, et son incidence continue d'augmenter en Amérique du Nord. La chimioembolisation trans-artérielle (TACE) est un traitement accepté pour le cancer du foie non chirurgical. Récemment, l'IRM de flux à quatre dimensions (4D) est devenue un outil important pour étudier la vitesse et le flux sanguin dans différents territoires vasculaires et permet une quantification a posteriori du flux sanguin dans un champ de vision 3D. L'analyse IRM en flux 4D pourrait ainsi être un avantage potentiel pour compléter l'évaluation pré-TACE des patients atteints de CHC non éligibles à la chirurgie et améliorer l'orientation des traitements endovasculaires.
L'objectif est d'étudier et d'évaluer la faisabilité des mesures d'IRM en flux 4D dans les artères hépatiques de patients atteints de carcinome hépatocellulaire (CHC) avant et après un traitement de chimioembolisation transartérielle (TACE).


